Nowości w Mnova 11
MNOVA 11.0.0 zawiera wiele nowych funkcji w modułach NMR, MS, Molecular Editor, Database, Verify i NMRPredict. Ponadto dodany został zintegrowany moduł PhysChem, jak również ważne ulepszenia w mechanizmie skryptowym.




logP
Molecular Volume,
Contact Surface Polar HBD,
Contact Surface Polar HBA,
Contact Surface Neutral,
Contact Surface Total,
logS,
Abraham A,
Abraham B,
Abraham E,
Abraham S,
Abraham V,
Boiling Point,
Henry’s Law Constant,
Hydroxy Radical Attack,
Melting Point,
Vapor Pressure,
logD,
Human Serum Albumin Affinity Constant,
Alpha Acid Glycoprotein Affinity Constant,
Membrane Affinity Constant,
Per cent Plasma Protein Binding,
Free Plasma Fraction,
Blood-Brain-Partition Coefficient,
Free Brain Fraction

Główne nowe funkcje oraz ulepszenia:
- znaczna poprawa jakości w rozwiązywaniu zadań i interpretacji widm 1D i 2D
- asystent zadań
- strzałki korelacji w cząsteczkach
- ulepszony workflow
- „jakość” przyporządkowania przedstawiona jest w tabeli przyporządkowań
- sugerowane „najlepsze przewidywanie widma NMR”
- moduł właściwości fizykochemicznych (PhysChem)
- ulepszoną obsługę eksperymentów NUS
- zaawansowane narzędzie dekonwolucji
- przetwarzanie DOSY
- nowy algorytm przetwarzania DOSY
- korekcja Non-Uniform Gradient (NUG)
- automatyczne śledzenie pików w Reaction Monitoring
- wsparcie technik kowariancji oraz spektroskopii Pure Shift w zaawansowanym przetwarzaniu widma NMR
- okienko typu Popup dla molekuł
- obliczanie jednorodności (“czystości”) pików w Mnova MS

Nowości w module Mnova NMR
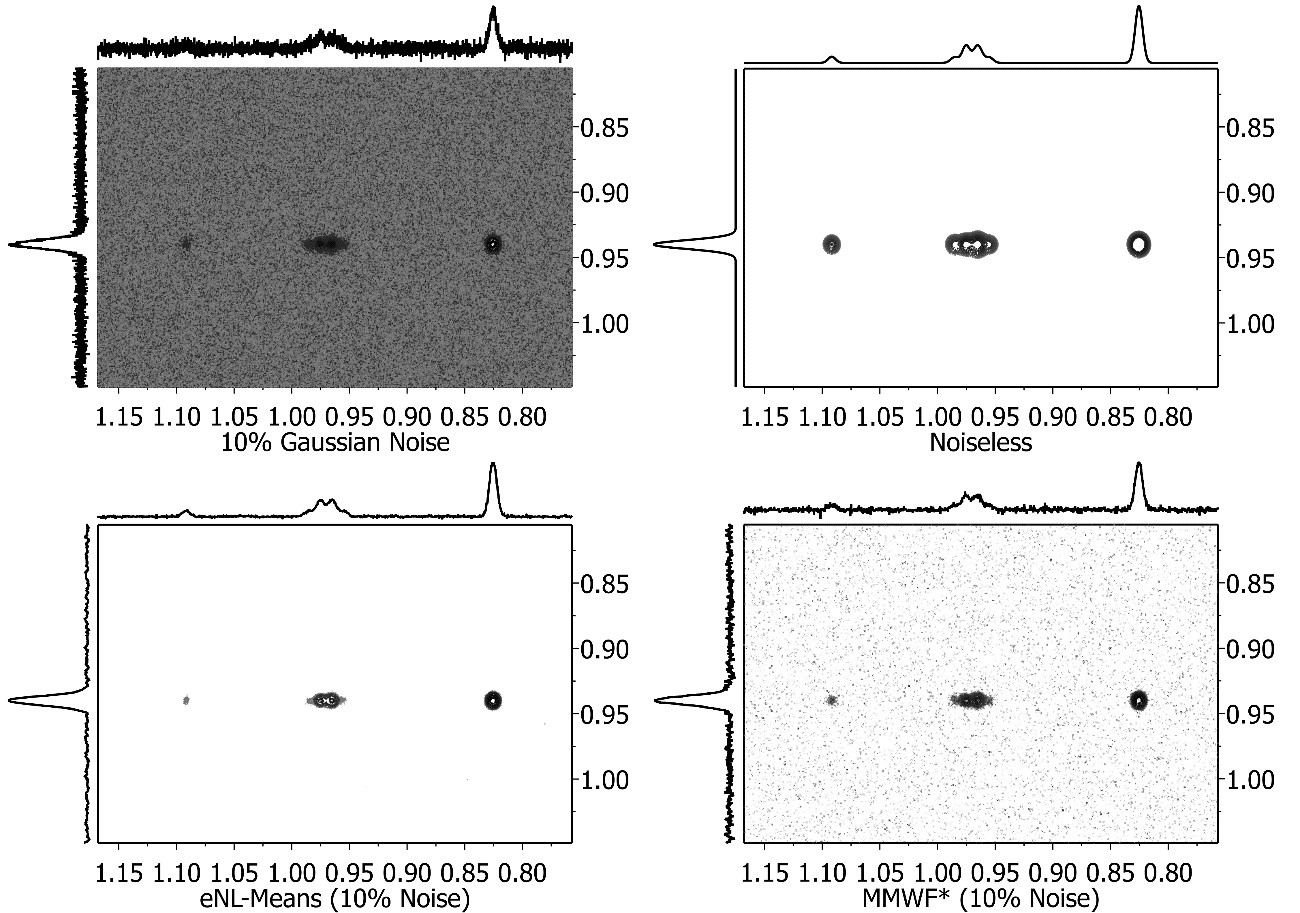
- Redukcja szumów
W dowolnej metodzie eksperymentalnej, rozróżnienie pomiędzy sygnałem i szumem ma ogromne znaczenie, szczególnie w przypadku widm NMR gdzie mamy do czynienia z ich niskią czułością eksperymentu. Jednym ze sposobów rozwiązania problemu jest stosowanie tak zwanych filtrów przestrzennych, również dla zbiorów danych 2D NMR. Mnova zawiera szereg filtrów mających na celu obniżenie poziomu szumu przy zachowaniu oryginalnej rozdzielczości widma
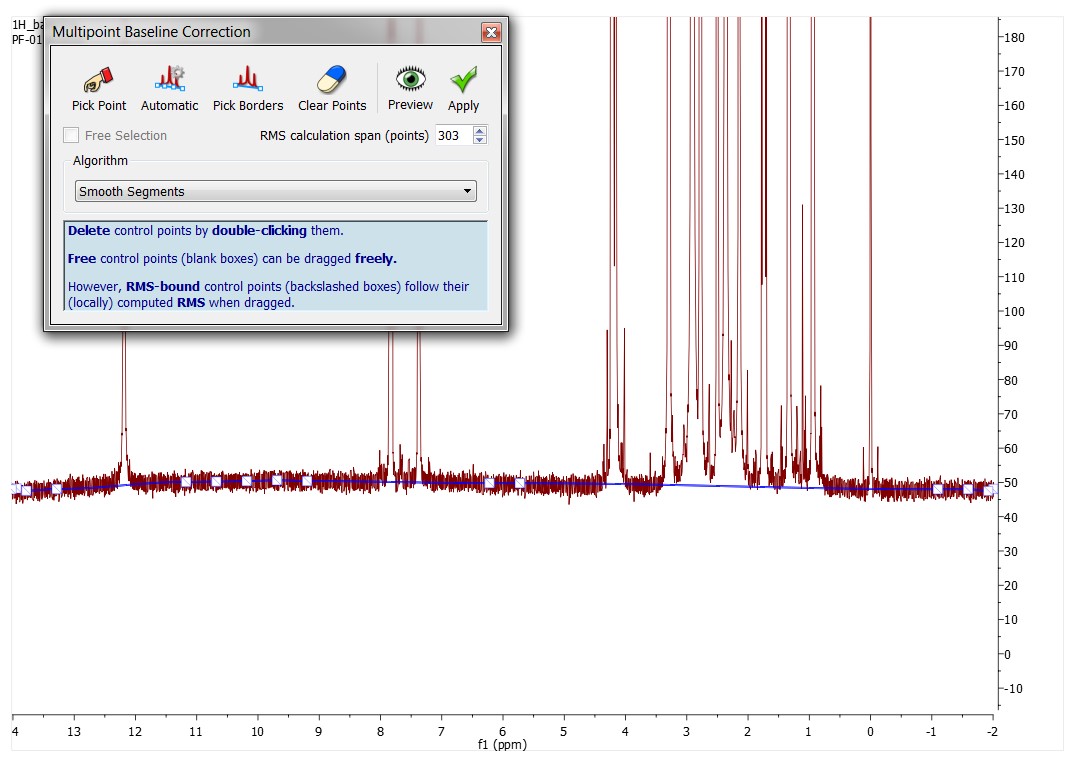
- Zastosowanie algorytmu GSD z wielopunktową korekcją linii bazowej
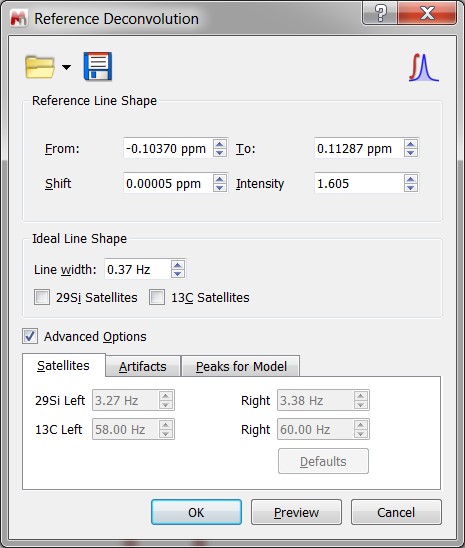
- Ulepszony moduł zaawansowanej dekonwolucji widma
- Ulepszona grupowa korekcja opóźnienia widma – zastosowanie tej metody pozwala prawidłowo obsługiwać wartości fazowe importowane z spektrometru
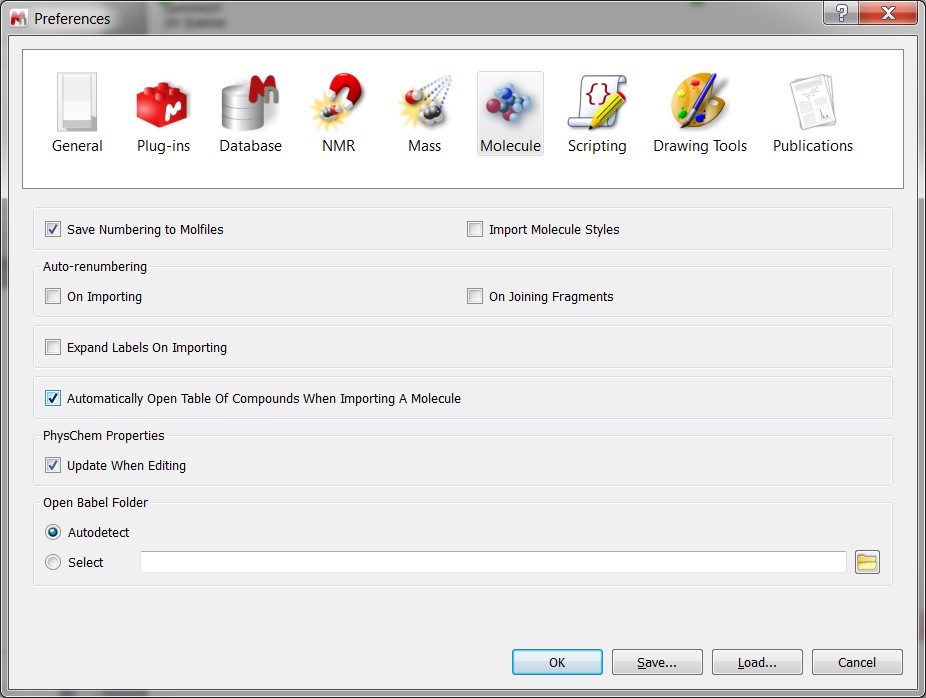
- Nowe okienko typu pop-up z ustawieniami wstępnymi i preferencjami przy otwieraniu każdej nowej cząsteczki
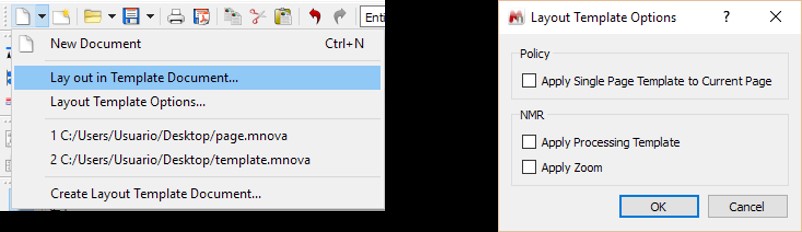
- Możliwość zastosowania szablonów do konkretnych stron dokumentu
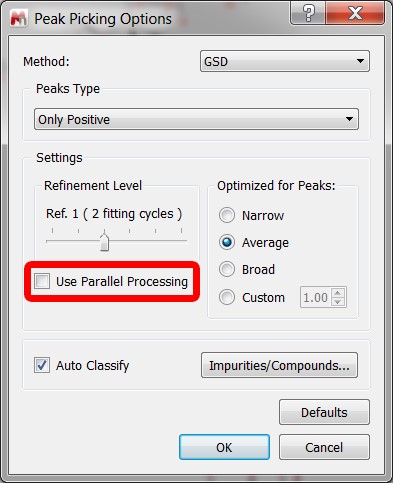
- Zrównoleglenie obliczeń dekonwolucji widma NMR
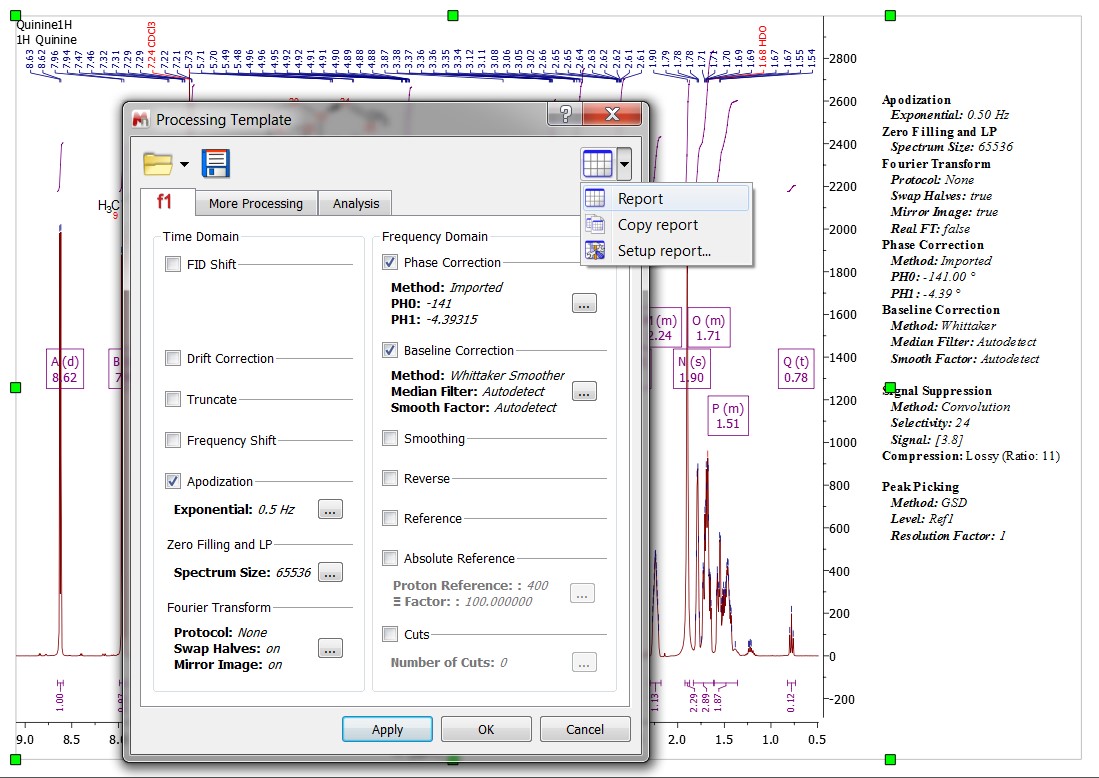
- Możliwość sporządzania raportów szablonów przetwarzania
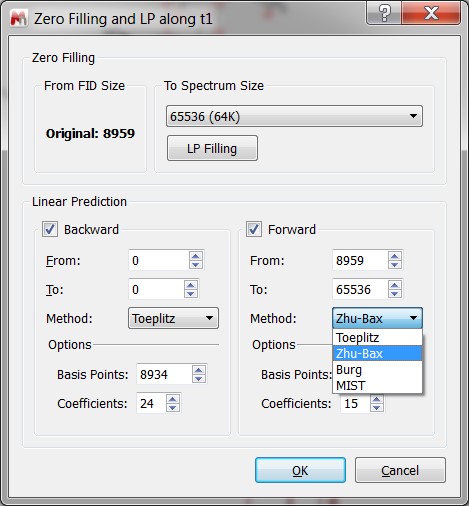
- Poprawione okno dialogowe dla technik depełniania zerami (Zero Filling) oraz liniowej predykcji (LP)
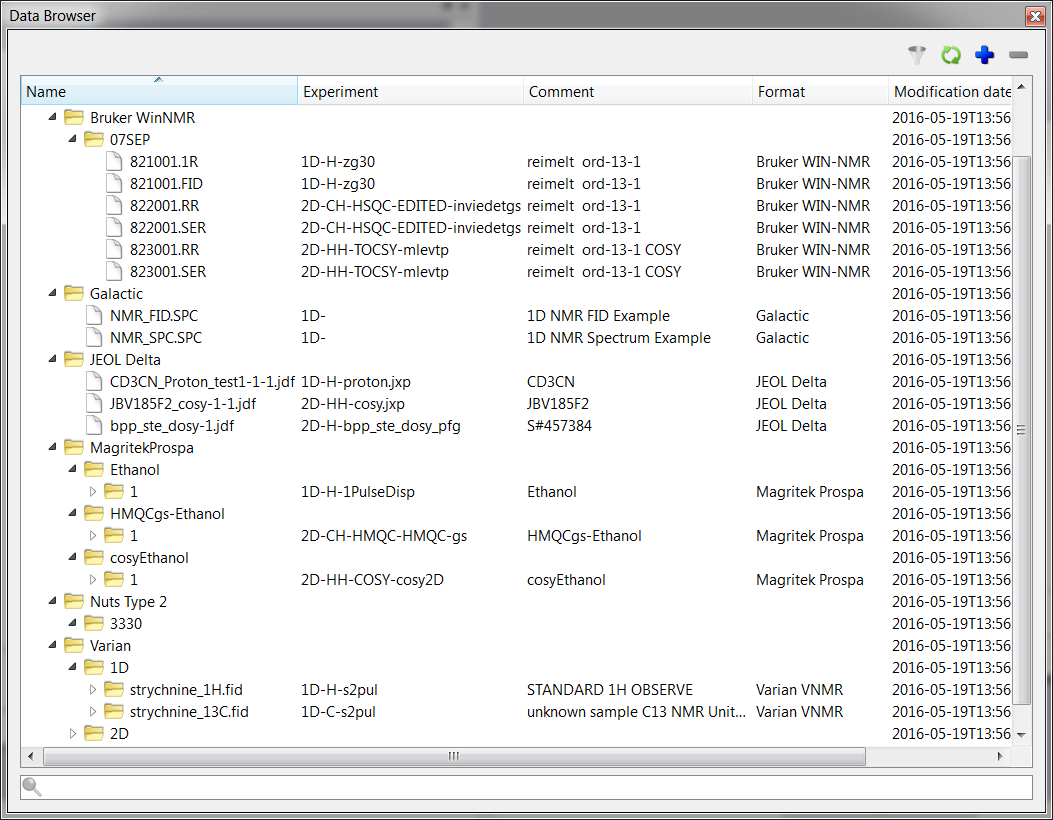
- Prezentowanie danych w oknie w rozróżnieniu po rodzaju eksperymentu
- Automatyczna aktualizacja śladów widm 1D i 2D po zastosowaniu skryptu Absolute Reference Script
- Możliwość wyboru koloru integralnych regionów wykresu w analizie danych
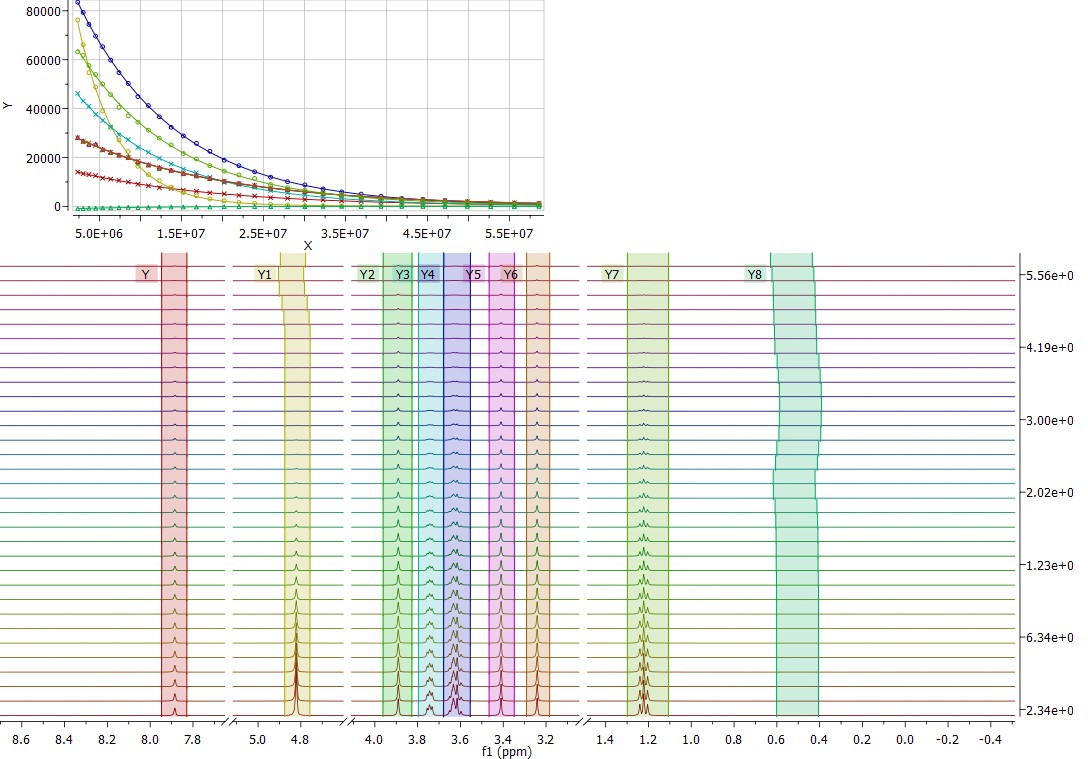
- Automatyczne śledzenie pików w analizie danych
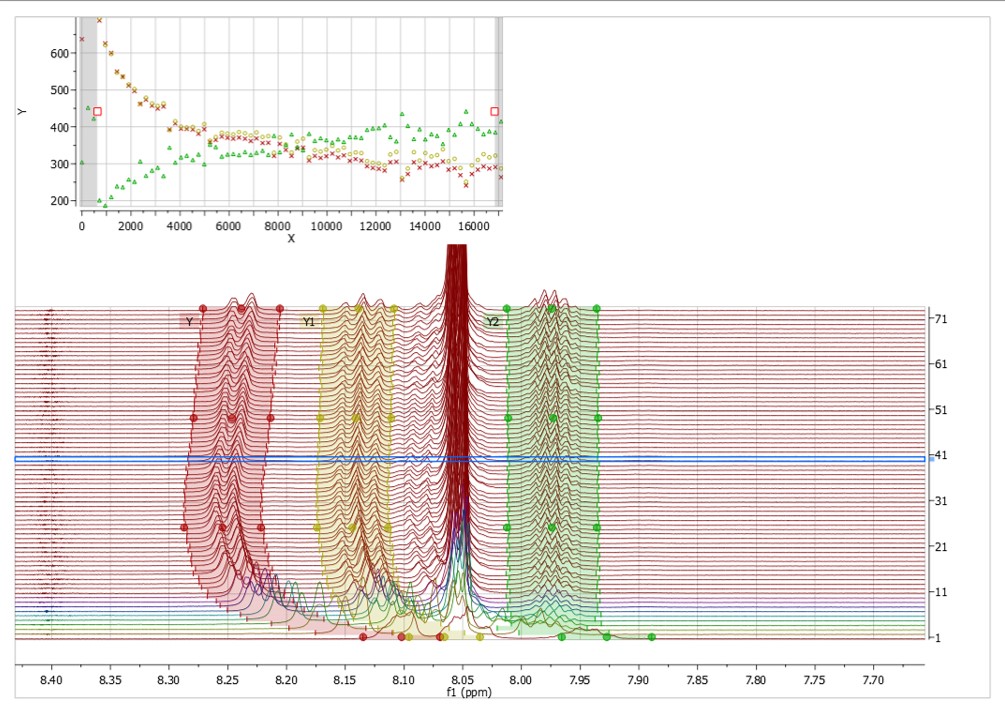
- Korekcja dla gradientu nieliniowości w przetwarzaniu DOSY
Nierównomierna korekta gradientu (NUG) może być stosowana przez zaznaczenie funkcji "Correct for non-uniform gradients " w oknie dialogowym przetwarzania DOSY - Możliwość otwarcia plików o formacie CNMR JCAMP-DX z poziomu Wiley Online Library
- Automatyczne dopasowanie widma do najlepszego związku po zastosowaniu skryptu TracesImported
- Import większej ilości parametrów z plików o formacie Brucker TopSpin JCAMP
- Przetwarzanie plików o formacie NUTS Type 1 pochodzących z oprogramowania Oxford Instruments
- Wsparcie dla widm NUS w formacie JEOL
- Kompatybilność techniki „przeciągnij i upuść” z nowym formatem danych VNMRJ3.2
- Rekonstrukcja sygnału FID

Nowości w module Mnova NMR Predict
- ulepszona dokładność predykcji
- możliwość wyboru domyślnego rozpuszczalnika w odniesieniu do prognoz widm 19F i 15N NMR
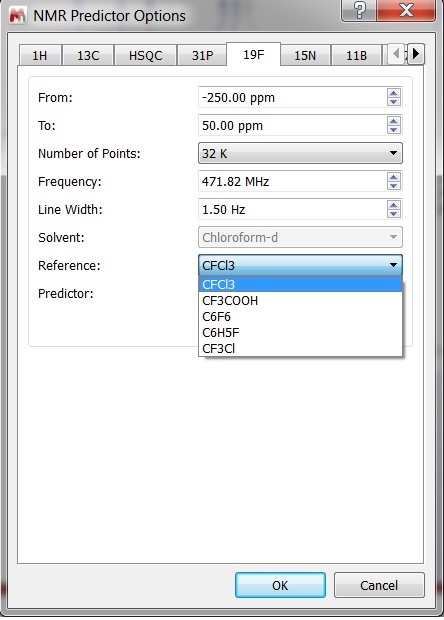
- możliwość aktualizacji przewidywanych widm na wykresach - przewidywane widma mogą być modyfikowane graficznie lub poprzez edycję wartości w tabeli Prediction lub Spin Simulation
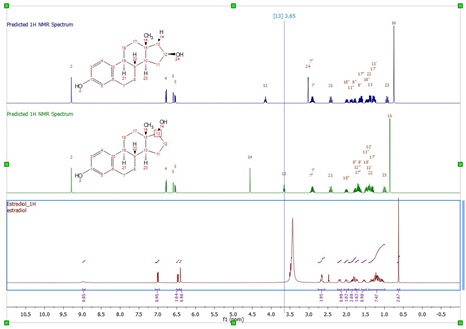
- możliwość do podkreślania w funkcji Predict & Highlight
- możliwość obliczeń stałej sprzężenia 3J HNCH
Molecule Editior
- pokazuje wektory do korelacji HMBC z nukleonami (15N, 29Si, itd.)
- struktury chemiczne w okienkach typu pop-up
- funkcja PhysChem w kolumnach
- możliwość obracania fragmentów cząsteczki
- zapisywanie danych predykcji po edycji cząsteczki za pomocą komendy Normalize Explicit Hydrogens Numbering
- zapisywanie danych predykcji po edycji cząsteczki za pomocą komend: Normalize Numbering, Concatenate Numbering, Add/Remove/Normalize Explicit Hydrogens
- funkcja wyświetlania strzałek korelacji poprzez umieszczenie wskaźnika myszy nad każdym atomem
- zmiana koloru tła przy wyborze komórki w tabeli związków
- możliwość zmiany rozmiaru czcionki liczb atomowych cząsteczki w tabeli związków
- funkcja obracania
- funkcja Lustra (góra-dół, lewo-prawo)
- możliwość edycji cząsteczki z tabeli związków
- możliwość sferycznego rysowania
- automatyczne otwieranie tabeli związków po imporcie cząsteczki
- odczyt parametrów z cząsteczki JCAMP

Nowości w module Mnova Verify
- funkcja dodawania flagi do definicji i pól w bazie danych
Teraz możliwe jest ustawiona flagi jako "Edytowalny", "Wymagany" lub "Unikalny":
- "Edytowalny": zestaw zapisany jako edytowalny będzie można edytować po przechowywaniu w bazie danych
- "Wymagany": zestaw pola jako wymagany będzie musiał posiadać wartości aby móc zostać zapisany do bazy danych
- "Unikalny": zestaw zapisany jako unikalny będzie wykorzystany w celu sprawdzenia zduplikowanych rekordów podczas zapisywania
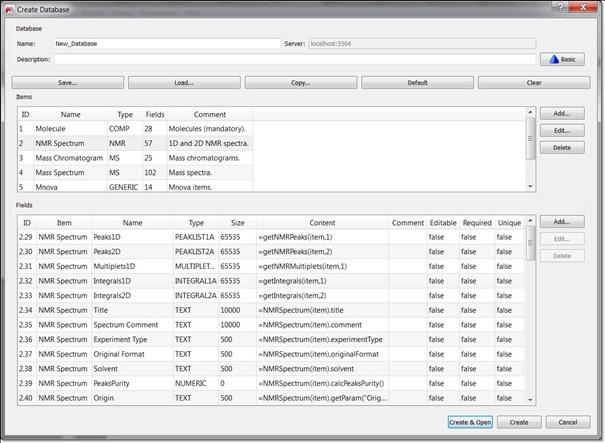
- Port Property w DVClient
- zawijanie tekstów podczas wyświetlania długich tekstów
- auto-skalowanie wysokości wiersza do zawartości pola
- przykładowe skrypty do wyszukiwania pików i multipletów i DB
- możliwość przechowywania chromatogramów UV/DAD i ELSD

Nowości w NPhysChem
Przewidywanie struktury z programem Mnova dostępne jest teraz na wyciągnięcie ręki. Moduł został opracowany we współpracy z Origenis GmbH, aby udostępnić skonsolidowane modele dla chemików z obszaru Drug Discovery i spoza niego. Standardowe właściwości modułu to funkcje:logP
Molecular Volume,
Contact Surface Polar HBD,
Contact Surface Polar HBA,
Contact Surface Neutral,
Contact Surface Total,
logS,
Abraham A,
Abraham B,
Abraham E,
Abraham S,
Abraham V,
Boiling Point,
Henry’s Law Constant,
Hydroxy Radical Attack,
Melting Point,
Vapor Pressure,
logD,
Human Serum Albumin Affinity Constant,
Alpha Acid Glycoprotein Affinity Constant,
Membrane Affinity Constant,
Per cent Plasma Protein Binding,
Free Plasma Fraction,
Blood-Brain-Partition Coefficient,
Free Brain Fraction

Nowości w module Mnova MS
- możliwość pobrania/zapisania ‘Peak Detection options’
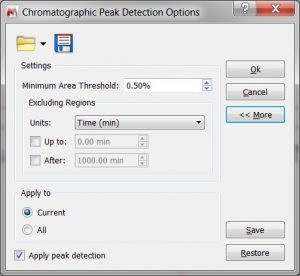
- możliwość określenia żądanej rozdzielczości
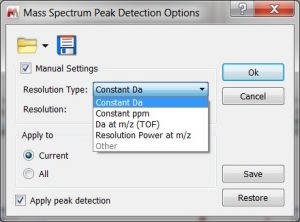
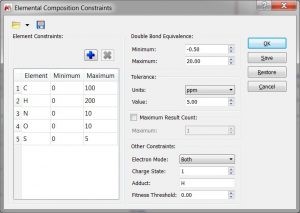
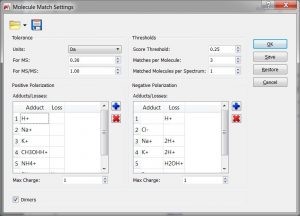
- funkcja pobierania/ zapisywania Elemental Composition i Molecular Match
- automatyczne wyrównanie śladów z zależności od maszyny MCMS
Funkcja ta jest przydatna, aby wyrównać ślady UV / DAD i MS stosując wartość korekcji
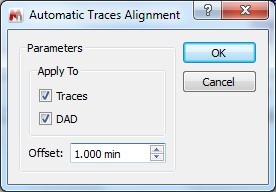
Parametry te mogą być predefiniowane z menu: ‘Edit/Preferences/Mass/Import’
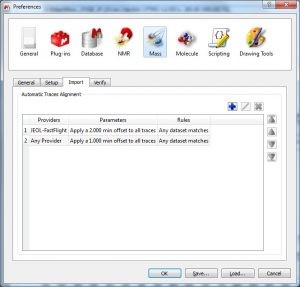
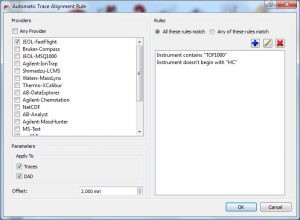
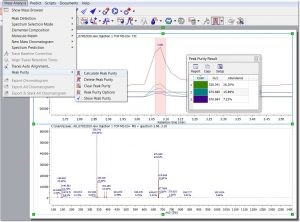
- obliczanie czystości pików
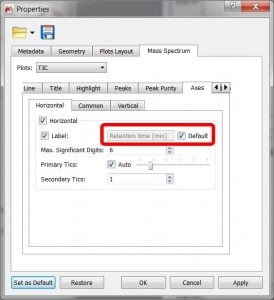
- udoskonalona etykieta osi pionowej na chromatografie
- nowy konwerter danych Advion MS