Mnova Binding

Wydajne narzędzie automatyzujące procesowanie danych spektroskopowych typu 2D HSQC (Heteronuclear Single Quantum Coherence) oraz obliczające wartości stałych dysocjacji Kd dla zestawu pików. Przeprowadzając analizy statystyczne Kd dla pików wielokrotnych można równie wysyłać zmierzone CSP do innych aplikacji (afinimetrów) dla zaawansowanej analizy z użyciem różnych powiązanych modeli.
Możliwości modułu
- Intuicyjny przepływ pracy pozwalający na analizę perturbacji przesunięć chemicznych CSP w sposób interaktywny i w pełni automatyczny
- Automatyczne śledzenie przesunięć pików oraz pomiar CSP, dopasowanie do koncentracji ligandów w celu obliczenia wartości stałych dysocjacji
- Narzędzia przetwarzania i układania widm 2D HSQC. Są one wyświetlane w różnych kolorach
- Korekta automatycznego śledzenia przesunięć pików w zagęszczonych obszarach
- Prosty eksport danych CSP do dalszych analiz
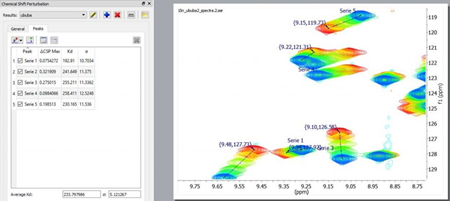
Śledzenie pików
Skumulowane widma HSQC z CSP są automatycznie śledzone i mierzone. Rysunek po lewej pokazuje ruch dwóch przypisanych pików. Okno dialogowe po prawej pokazuje szczegóły jednego ze śledzonych pików "13C-H", w tym stosunek stężeń liganda / białka, zmierzone CSP w dowolnym wymiarze, znormalizowanie CSP i dopasowanie wyników Kd i błędu.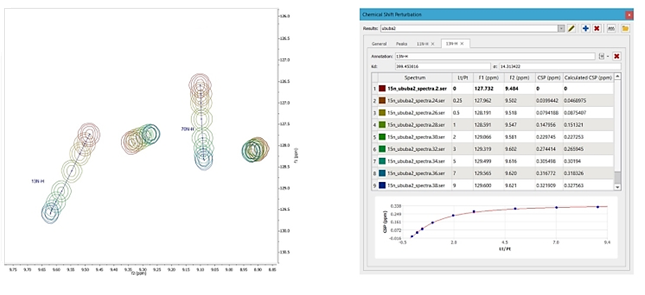
Pomiar i dopasowanie
Zakładka ogólna „Chemical Shift Perturbation” pokazuje listę schematów kolorów dla widm miareczkowania, stężeń białka, stężeń ligandów i stosunku stężenia ligandu do białka. Pokazuje również listę pomiarów CSP i dopasowanie Kd dla wszystkich analizowanych pików, a także średni i standardowy błąd Kd.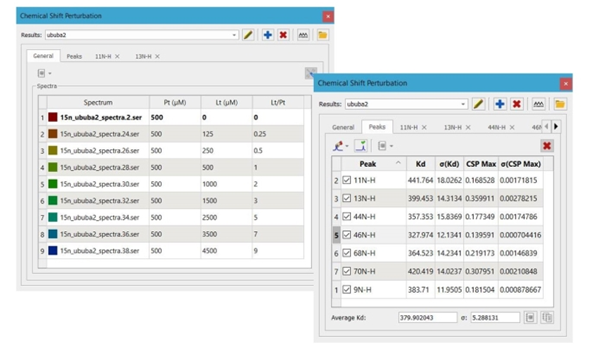
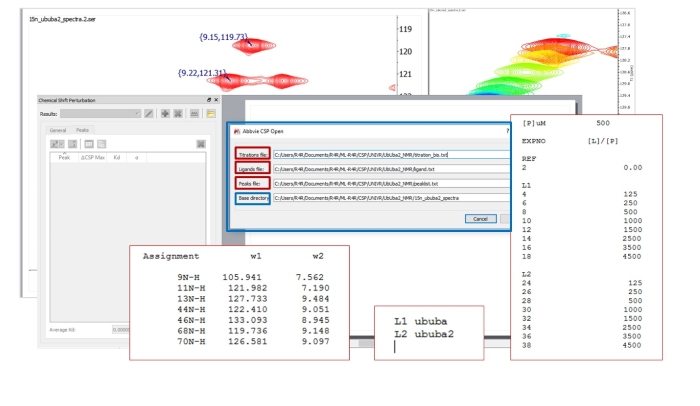
W pełni automatyczne analizy
Seria miareczkowań może być określona przez ich umieszczenie po kolei w "pliku miareczkowania". Nazwy ligandów są kodowane w "pliku ligandów". Gdy CSP automatycznie uruchamia analizę, tworzy różne pliki Mnova dla każdego miareczkowania wymienionego w "pliku miareczkowania", a dokumenty Mnova są odpowiednio nazwane nazwami ligandów.