Nowości Mnova 14.3

1. Nowy produkt! Mnova Screen 2D
Ten produkt jest powiązany z Mnova Screen, który może być użyty do przetwarzania widm 1D NMR obserwowanych przez ligandy (STD, T1rho, CPMG, WaterLogsy, itp.). Screen 2D przetwarza obserwowane białka 2D 1H-15N, 1H-13C lub H1-13C/15N z podwójną korelacją heterojądrową (HSQC lub HMQC) w celu znalezienia wiążących ligandów w oparciu o zaburzenia przesunięcia chemicznego.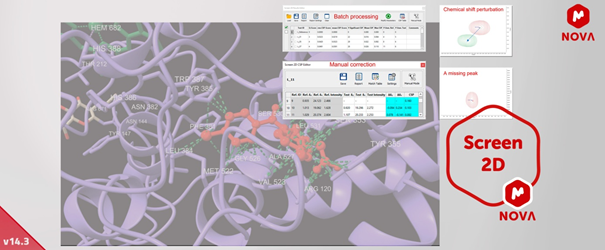
2. Obsługa plików Waters Empower i Thermo Chromeleon - MSChrom
Z przyjemnością informujemy, że Mnova MSChrom (dawniej Mnova MS) może teraz importować i obsługiwać pliki Water Empower i Thermo Chromeleon. To narzędzie zostało dodane do wstążki Mnova Tools w menu rozwijanym „Import”. Należy pamiętać, że to narzędzie musi mieć zainstalowane na tym samym komputerze co Empower i/lub Chromeleon.
To narzędzie działa w dwóch krokach: eksportuje dane z Empower lub Chromeleon do pliku, a następnie importuje ten plik do Mnova.
3. Wsparcie dla frakcji (do tej pory Waters MassLynx) - MSChrom
Wdrożono nowy konwerter do obsługi ułamków, który automatycznie importuje informacje o frakcjach z pliku podczas ładowania pliku lub folderu MS. W ten sposób można ustawić nazwę pliku frakcji według dostawcy w arkuszu Fraction sheet of the Mass Preferences import sheet. Można także zdefiniować reguły, aby ustawić nazwę pliku frakcji w funkcji metadanych MS. Należy pamiętać, że na tym etapie obsługiwane są tylko informacje o frakcjach Waters-MassLynx, ale wkrótce wsparcie zostanie rozszerzone na inne frakcje.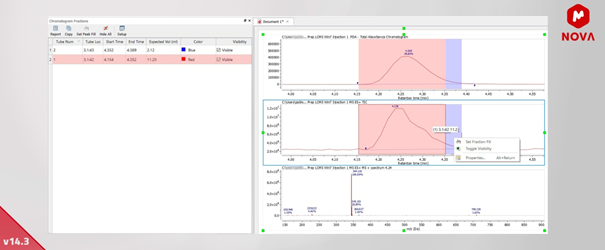
Informacje z pliku frakcji są pokazane w tabeli Chromatogram Fractions. Każdej frakcji automatycznie przypisywany jest kolor.
4. Zapisz cały dokument jako JCAMP - Mnova General
Z przyjemnością informujemy ulepszeniu sposobu obsługi plików JCAMP. Wdrożono nowy filtr plików „Dokument JCAMP-DX” (*.jdx *.dx *.jcm *.cs *.jcamp), który jest analogiczny do pozostałych JCAMP-DX z tym wyjątkiem, że podczas zapisywania, zapisywany jest cały dokument.
Ten konwerter będzie zawsze włączony, bez względu na dokonany wybór. Zasadniczo można zapisać cały dokument z kilkoma stronami jednocześnie jako plik JCAMP.
5. Ulepszony eksport JCAMP - NMR
Udoskonalono szereg funkcji zapisywania oryginalnych ustawień podczas eksportowania do pliku JCAMP, takich jak:- 1D or 2D cuts
- Zoom
- Stack mode
- Traces in the spectru
6. Wiele cząsteczek w jednym rekordzie DB - Mnova DB
Mnova DB pozwala teraz na zapisanie kilku molekuł w jednym rekordzie i tworzenie baz danych bez molekuł. (Metoda Molecule nie jest już obowiązkowa dla nowej bazy danych).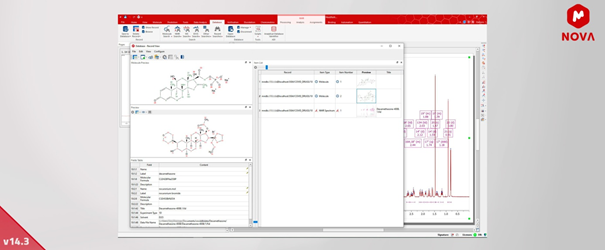
7. Nowy tryb niestandardowy do wybierania widm MS z chromatogramów – MSChrom – wprowadzono nowy tryb celownika „Customized Peak”.
Ten tryb może być użyty do wybrania danego piku na chromatogramie, zintegrowania procentu skanów MS tego piku i automatycznego odejmowania skanów przed/po tym piku. Implementacja zapewnia, że integracja posiada co najmniej jeden skan MS. Ten tryb został dodany do menu Tools > Crosshair/Select w sekcji Mass. Zaimplementowano również odpowiednią funkcję skryptową.
Można także zmienić ustawienia za pomocą menu rozwijanego „Customized Peak Settings”. W ten sposób można zmodyfikować procent całkowania i wybrać całkowanie piku odniesienia.
8. Obliczenia przydatności systemu dla danych chromatograficznych - MSChrom
Zaimplementowano obliczenia przydatności systemu (width, resolution, number of theoretical plates, tailing factor and signal-to-noise) we wtyczce MSChrom.Pierwszy z nich pokaże szerokość każdego piku przy określonym procencie wysokości. Pozostałe cztery obliczenia pokażą odpowiednie obliczenia przydatności systemu dla każdego piku.

Nowy przycisk „System Suit” został dodany do paska narzędzi tabeli Chrom Peaks. Istnieją dwa predefiniowane ustawienia, USP i EP, które zawierają wartości odpowiednich parametrów odpowiednio dla Farmakopei amerykańskiej i europejskiej.
9. SIMCA Model Classification – Chemometrics
Miękkie niezależne modelowanie przez analogię klas (SIMCA) to statystyczna metoda nadzorowanej klasyfikacji danych.W celu zbudowania modeli klasyfikacyjnych, próbki należące do każdej klasy muszą być analizowane za pomocą analizy głównych składowych (PCA), z której zachowane są tylko istotne składowe.
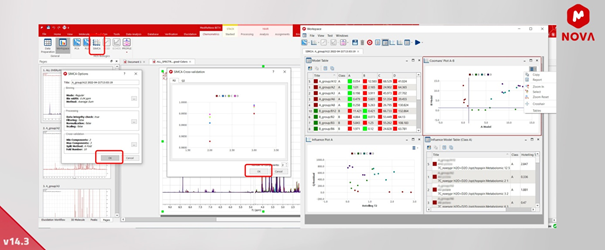
Uruchamianie analizy SIMCA jest teraz możliwe za pomocą zakładki Chemometrics. Początkowo przeprowadzana jest walidacja krzyżowa (dostępne są te same ustawienia, które są używane do analizy PCA). Panel SIMCA pokazuje wynikową tabelę modelu. Można również uzyskać dodatkowe wykresy, wykresy wpływów (dla każdej klasy) i wykres Coomana (dla każdej pary klas).
10. Ulepszenia w skalach wykresów – MSChrom
Wprowadzono synchronizację czasu retencji chromatogramów MS, a także dodano synchronizację skali m/z widm masowych.Można włączyć te funkcje domyślnie w Mnova > Preferences > Mass > General > Scales, a także sterować synchronizacją za pomocą przycisku w menu Mass > Tools in the ribbon.
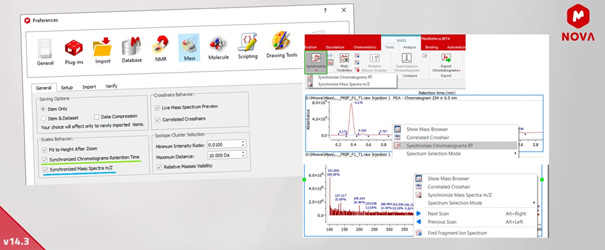
Ponadto poprawiono sposób synchronizowania śladów podczas powiększania, nawet jeśli ślady nie zawierają danych w określonym zakresie, wyświetlając pusty region.
11. Nowa interaktywna przeglądarka baz danych - Mnova DB
Wygenerowano nową interaktywną przeglądarkę baz danych, która pomoże Ci w zapytaniach do bazy danych.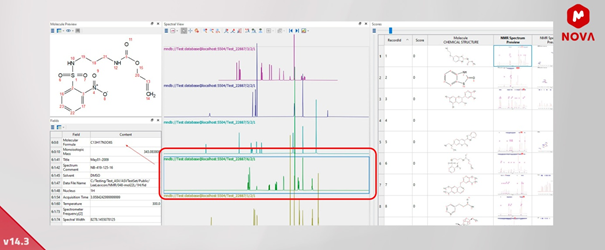
To narzędzie zawiera widżet „widoku widmowego”, który pomaga w interakcji z pojedynczymi widmami lub w trybie stosowym w Mnova DB bez konieczności kopiowania zapytań do samej Mnova.