Nowości Mnova 15
1. Mnova NMR
Ulepszony moduł DOSYMnova 15 wprowadza przemyślane ulepszenia modułu DOSY. Dzięki tej aktualizacji udoskonalone zostały algorytmy w celu jeszcze dokładniejszego obliczania widm DOSY, integrując ulepszoną analizę błędów i dopasowanie Monte Carlo. Oprócz tych ulepszeń technicznych obejmuje rozszerzoną obsługę szerszej gamy eksperymentów PFG. Częścią tej aktualizacji jest również wprowadzenie nowej funkcji graficznej, opracowanej w celu zapewnienia użytkownikom wyraźniejszej i bardziej szczegółowej analizy wizualnej wyników.
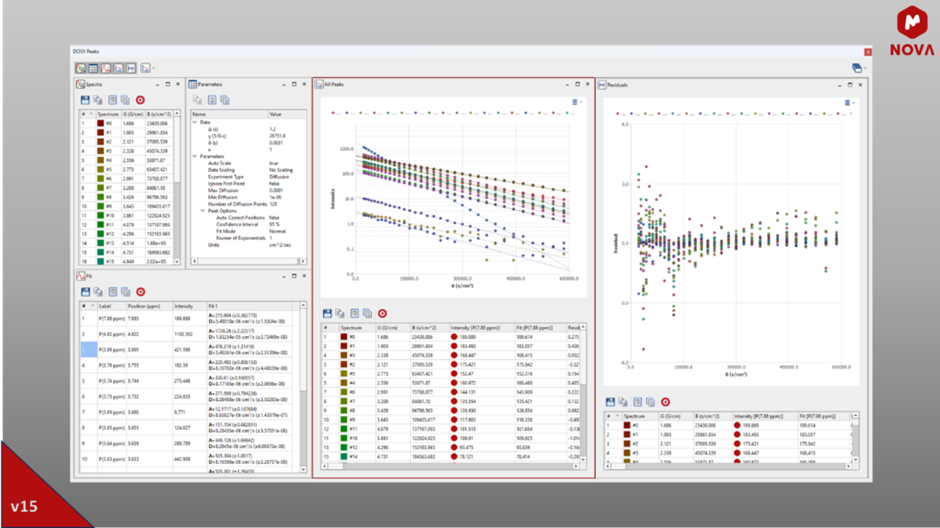
Rys. 1: Nowy panel DOSY Peaks zapewnia obszerną analizę wartości dyfuzji wraz z interaktywnymi wykresami.
Możliwości przetwarzania danych 3D NMR
Mnova NMR poszerzyła swoje horyzonty dzięki nowatorskiej obsłudze zbiorów danych 3D NMR, zaczynając od plików przetworzonych przez Brukera (np. w formacie 3rrr). Producent aktywnie rozszerza możliwości, aby uwzględnić dane innych formatów i spodziewamy się wkrótce zaoferować Państwu to ulepszenie. Co więcej, zaktualizowana wersja zapewnia bezproblemową integrację z listami pików z oprogramowania NMR, takiego jak CARA, CCPN, Sparky i TopSpin, ułatwiając badaczom płynniejszą pracę.
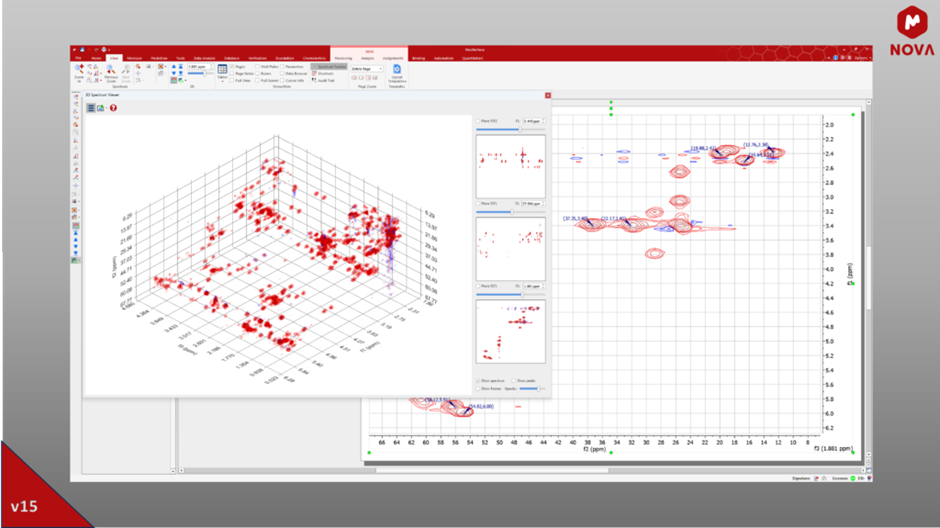
Rys. 2: Mnova 15 obsługuje teraz eksperymenty przetwarzane 3D NMR.
Nowa przeglądarka widma 3D
Zaawansowana przeglądarka 3D NMR, początkowo zaprojektowana do analizy widmowej 3D, teraz służy również do wizualizacji konwencjonalnych eksperymentów 2D i eksperymentów tablicowych NMR (tj. pseudo 2D), takich jak DOSY, zapewniając użytkownikom dynamiczne i wzbogacające wrażenia z eksploracji wizualnej.
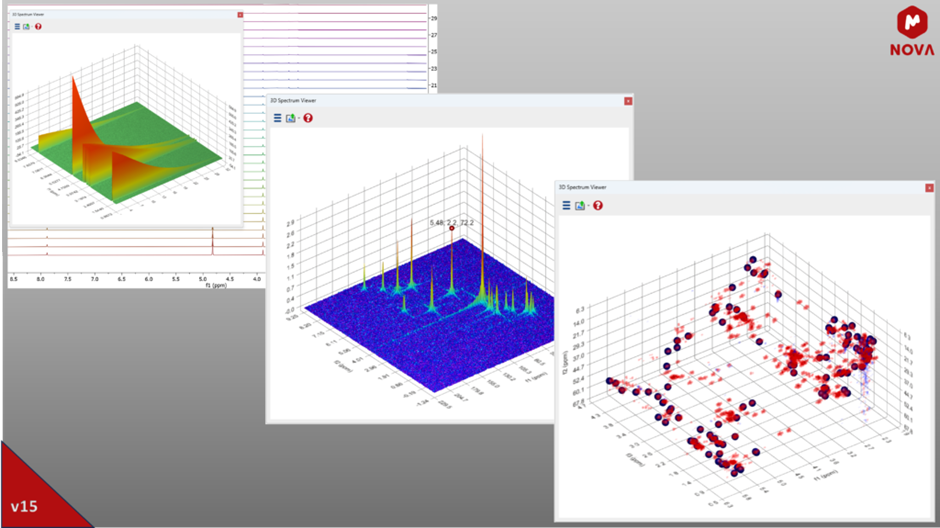
Rys. 3: Nowej przeglądarki 3D można używać do wyświetlania eksperymentów pseudo 2D NMR wraz z tradycyjnym wyświetlaniem widm skumulowanych, widm 2D i pików 3D NMR.
Algorytm wybierania wartości szczytowych oparty na sztucznej inteligencji
We współpracy z firmą Bruker powstał zaawansowany dodatek do Mnova NMR: algorytm wybierania szczytów oparty na głębokim uczeniu (1). To najnowocześniejsze narzędzie zwiększa możliwości zaufanego algorytmu GSD (Global Spectral Deconvolution), szczególnie w uzyskanie dokładnych ilościowych wyników NMR (qNMR). W połączeniu z nowatorską, pionierską, obsługiwaną przez sztuczną inteligencję automatyczną korekcją linii bazowej i fazy (patrz następny punkt), wyznacza nowy standard dokładności i wydajności operacyjnej w analizie widmowej NMR.
(1) Schmid, N., Bruderer, S., Paruzzo, F., Fischetti, G., Toscano, G., Graf, D., Fey, M., Henrici, A., Ziebart, V., Heitmann, B., Grabner, H., Wegner, J. D., Sigel, R. K. O. i Wilhelm, D. (2023). Dekonwolucja widm 1D NMR: podejście oparte na głębokim uczeniu się. W Journal of Magnetic Resonance (tom 347, s. 107357). Elsevier BV. https://doi.org/10.1016/j.jmr.2022.107357
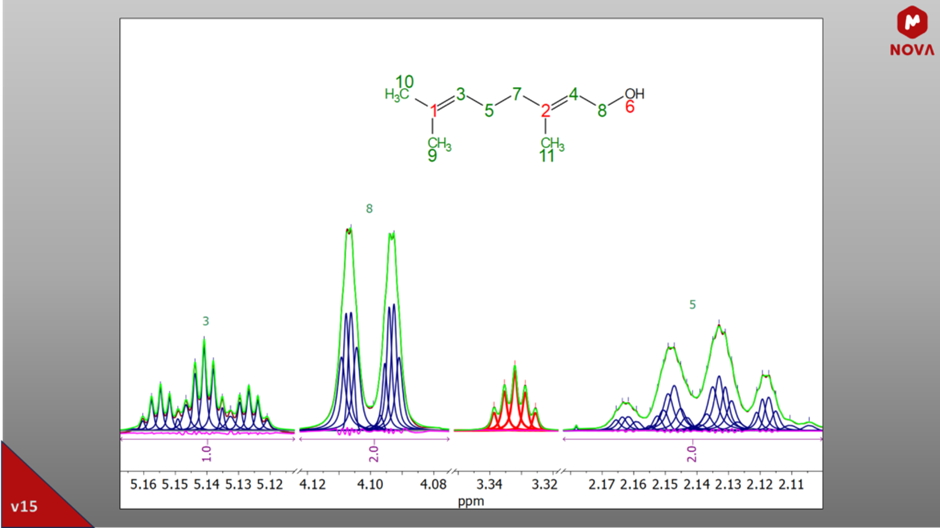
Rys. 4: Pełna dekonwolucja widma 1H-NMR przy użyciu nowego algorytmu opartego na głębokim uczeniu się wykazuje wyjątkową wydajność. Na szczególną uwagę zasługują pozostałości zaznaczone kolorem fuksja. Rozplątane piki są reprezentowane przez krzywe niebieską (związek) i czerwoną (rozpuszczalnik), podczas gdy krzywa zielona odpowiada skumulowanej sumie wszystkich tych pików syntetycznych.
Zaawansowana korekcja fazy i linii bazowej
Wprowadzono nowatorski algorytm głębokiego uczenia się, zaprojektowany do jednoczesnej korekcji zniekształceń fazowych i linii bazowej – co stanowi bardziej zintegrowane rozwiązanie dla wszechobecnych wyzwań NMR. To osiągnięcie, będące kolejną udaną współpracą z firmą Bruker, jest początkowo dostępne wyłącznie dla widm 1H NMR. Zintegrowany algorytm stanowi doskonałe uzupełnienie algorytmu wybierania wartości szczytowych opartego na głębokim uczeniu się (patrz poprzedni punkt), usprawniając ogólną analizę widmową.
Ulepszone algorytmy korekcji linii bazowej
Uznając potrzebę szybkiego przetwarzania, Mnova NMR zawiera teraz ulepszone algorytmy korekcji linii bazowej, które poszerzają ich zastosowanie w różnych eksperymentach NMR. Ulepszenia te zapewniają harmonijną równowagę pomiędzy wydajnością i precyzją, spełniając szybko zmieniające się wymagania współczesnej analizy spektralnej.
2. Mnova MSChrom
Z dumą prezentujemy najnowsze zaawansowane funkcje MSChrom, rozwiązania LC/GC-MS, wcześniej nazywanego Mnova MS. Te innowacyjne udoskonalenia nie tylko podnoszą precyzję i wydajność wtyczki, ale także podkreślają zaangażowanie Mestrelab Research w zapewnianie użytkownikom ultranowoczesnych narzędzi do ich potrzeb analitycznych. Poznaj niezrównane możliwości MSChrom opisane poniżej:Teraz MSChrom zawiera nowy algorytm Enhanced Peak Picking Algorithm stanowiący alternatywę dla klasycznego algorytmu używanego w poprzednich wersjach MSChrom. W ramach ulepszonego algorytmu wybierania pików dostępne są nowe ustawienia umożliwiające odtworzenie najlepszych wyników w zakresie wykrywania i integracji, w tym korekcja linii bazowej, funkcje wygładzania i nowa funkcjonalność oparta na poziomach nasycenia, gdzie użytkownicy mogą ustawić próg obfitości w celu zidentyfikowania przeciążonych pików z powodu do nasycenia detektorów i dokonać na nich korekcji.
Przedstawiamy również nowe narzędzie „Blind Regions” – bardzo przydatny dodatek. Narzędzie to umożliwia selektywne wykluczanie niepożądanych regionów z chromatogramów podczas procesów analitycznych, takich jak wybieranie pików lub jakakolwiek ogólna obróbka danych. Obszary niewidome pozostaną ignorowane, co pozwoli zaoszczędzić czas i zasoby oraz zapewnić lepszą powtarzalność wyników. Jest to szczególnie przydatne w przypadkach, gdy pojawiają się wyzwania w określonych obszarach (front rozpuszczalnika w LC, krwawienie z kolumny w GC itp.) lub, na przykład, gdy decydujesz się na odrzucenie granic chromatogramu.
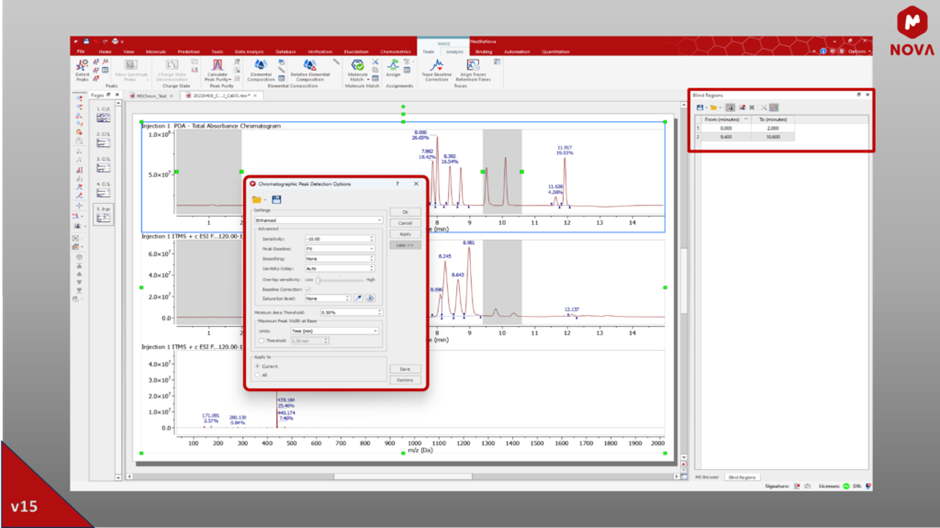
Rys. 5: Panel ustawień wykrywania pików chromatograficznych i tabela obszarów ślepych wyświetlana po procesie wybierania pików i zastosowaniu dwóch ślepych obszarów na chromatogramie.
Odejmowanie tła
Ta nowa funkcja ma na celu ułatwienie odejmowania tła w zbiorach danych, oferując możliwość zastosowania widm masowych i odejmowania UV w celu zmniejszenia zawartości zanieczyszczeń i zaszumionych wspólnych pików uzyskanych z systemu analitycznego itp. w celu uzyskania bardziej precyzyjnej analizy danych.
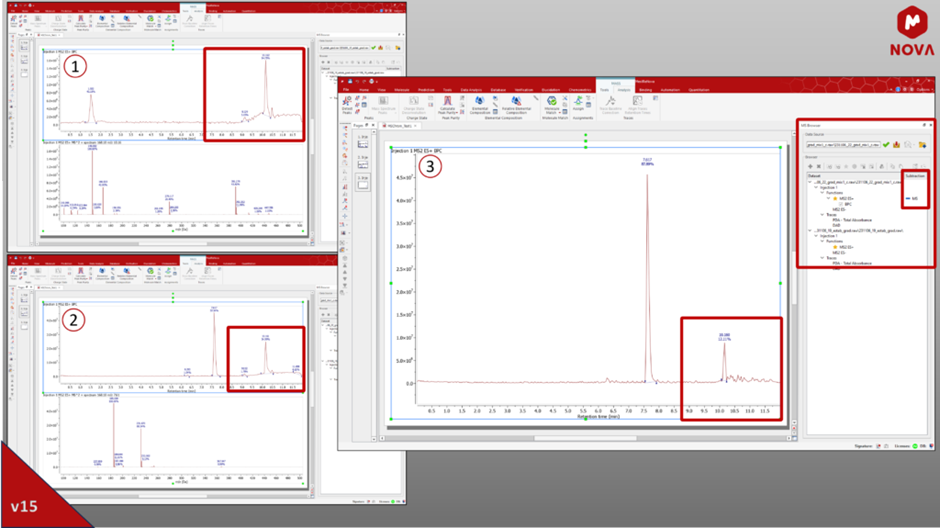
Rys. 6: Próbka nr 2 i próba ślepa nr 1 użyte do zastosowania odejmowania tła MS i wynik 3.
Obsługiwane formaty MSChrom
Przeglądaj listę dostawców danych uporządkowaną według systemu operacyjnego obsługiwanego przez Mnova MSChrom. Należy pamiętać, że Mnova 15 nie obsługuje już formatów Windows x32: https://resources.mestrelab.com/ms-supported-formats/
3. Chenometria
Aby zwiększyć dostępność i funkcjonalność analizy chemometrycznej w programie Mnova, z radością ogłaszamy dostępność trzech różnych pakietów chemometrycznych, każdy zaprojektowany w celu zaspokojenia różnych poziomów potrzeb użytkowników:- Mnova Basic Chenometrics - ten pakiet podstawowy oferuje podstawowe możliwości chemometrii, koncentrując się na najbardziej podstawowej funkcjonalności analizy głównych składowych (PCA). Jest to idealny punkt wyjścia dla użytkowników, którzy nie mają doświadczenia z chemometrią.
- Mnova Advanced Chemometrics - ten zaawansowany pakiet, oparty na pakiecie Basic, zawiera wszechstronne funkcje PCA i rozszerza swoje możliwości o dodatkowe algorytmy chemometrii, takie jak częściowe najmniejsze kwadraty (PLS) i miękkie niezależne modelowanie analogii klas (SIMCA). Ten pakiet jest przeznaczony dla użytkowników poszukujących bardziej wyrafinowanych narzędzi analitycznych, umożliwiających tworzenie, zapisywanie i wykonywanie modeli, wraz z ulepszonymi funkcjonalnościami zapewniającymi bardziej dogłębne doświadczenie chemometrii.
- BioHOS - jako najbardziej kompleksowy pakiet w naszej ofercie, MBioHOS obejmuje wszystkie funkcje pakietu Advanced Chemometrics i dodatkowo rozszerza jego zastosowania o specyficzne algorytmy do analizy NMR leków bioterapeutycznych, takich jak przeciwciała monoklonalne. Należą do nich algorytmy takie jak CCSD, ECHOS i PROFILE.
4. Mnova (generalnie)
Nowe narzędzie do rysowania peptydów - Biosequence ToolsDzięki narzędziom Biosequence użytkownicy Mnova mogą rysować łańcuchy aminokwasów, takie jak peptydy. To narzędzie to rozwiązanie oszczędzające czas, z przyjaznym dla użytkownika interfejsem, które zawiera pełną listę „standardowych” i „niestandardowych aminokwasów”, oferując możliwość wyboru pomiędzy kodami jedno- lub trzyliterowymi oraz konfiguracją L lub D. Po utworzeniu struktur peptydowych możesz rozszerzyć każdy aminokwas w łańcuchu, aby poprawić przestrzenne widzenie struktury, zmienić kod literowy, pobrać skompresowaną nazwę za pomocą narzędzia IUPAC Name i zapisać ją jako plik .mol.
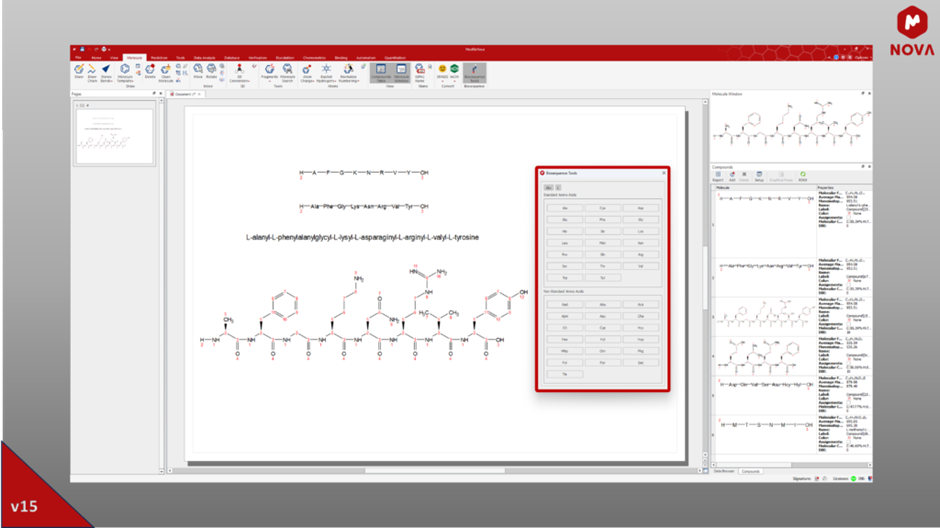
Rys. 7: Nowy panel Biosequence Tools zapewnia wiele opcji rysowania struktur peptydowych i łatwego manipulowania nimi.
5. Mnova ElViS (spektroskopia elektroniczna i wibracyjna)
Aby usprawnić przetwarzanie danych pochodzących z technik spektroskopii elektronicznej i spektroskopii wibracyjnej, Mnova 15 wyposaża oparte na PLS narzędzie do korekcji linii bazowej (IarPLS) w przycisk umożliwiający automatyczne obliczenie niemal optymalnej wartości parametru asymetrii. Dodatkowo zintegrowane zostały trzy nowe tryby przetwarzania normalizacyjnego, tj. największy szczyt, całka i długość wektora. Te dwie kluczowe funkcje przyczyniają się do znaczącej aktualizacji naszej wtyczki ElViS. Celem jest uproszczenie procesu korekty wartości bazowej, usprawnienie przepływu pracy i zapewnienie szybkiej metody normalizacji danych w celu rozwiązania problemów ilościowych.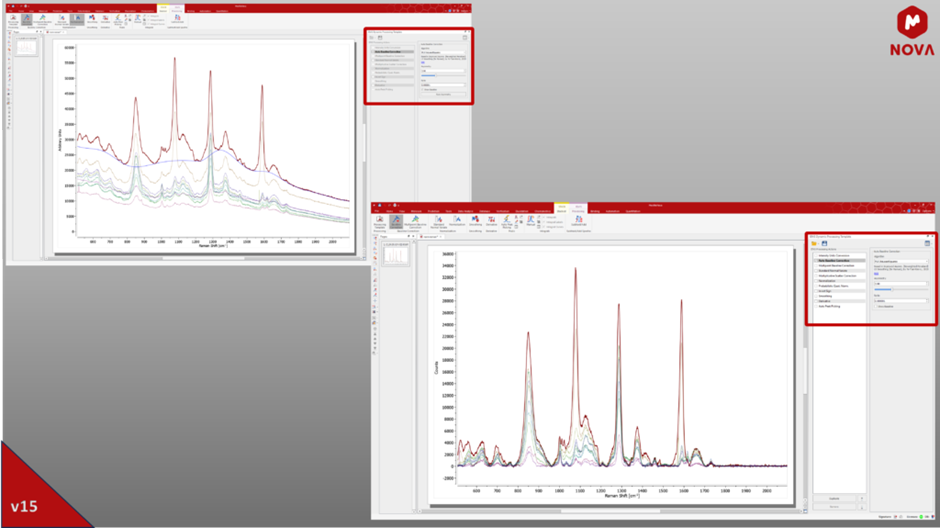
Rys. 8: Automatyczna korekta linii bazowej przy użyciu algorytmu IarPLS i wyświetlana opcja Show Baseline.
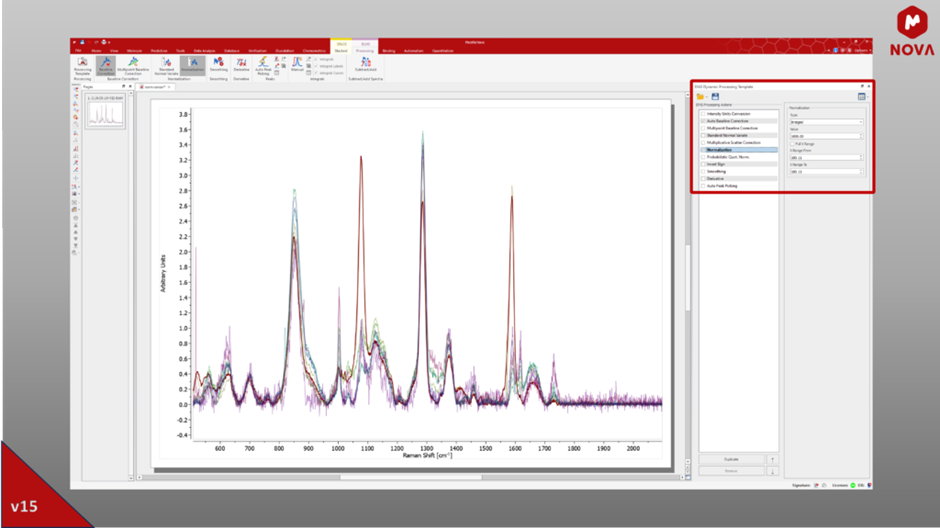
Rys. 9: Wynik normalizacji typu całkowego po korekcie linii bazowej opartej na IarPLS.
6. Mnova DB
Mnova 15 zawiera ulepszenia wtyczki DB, aby usprawnić tworzenie i zarządzanie bazami danych, zapewniając szereg kompleksowych opcji. Godne uwagi ulepszenia obejmują uwzględnienie stereochemii w zapytaniach o strukturę, „klikane” łącza w przeglądarce DB, dodano kolumny notacji SMILES i InChi, co przyczyniło się do bardziej wydajnego i przyjaznego dla użytkownika doświadczenia.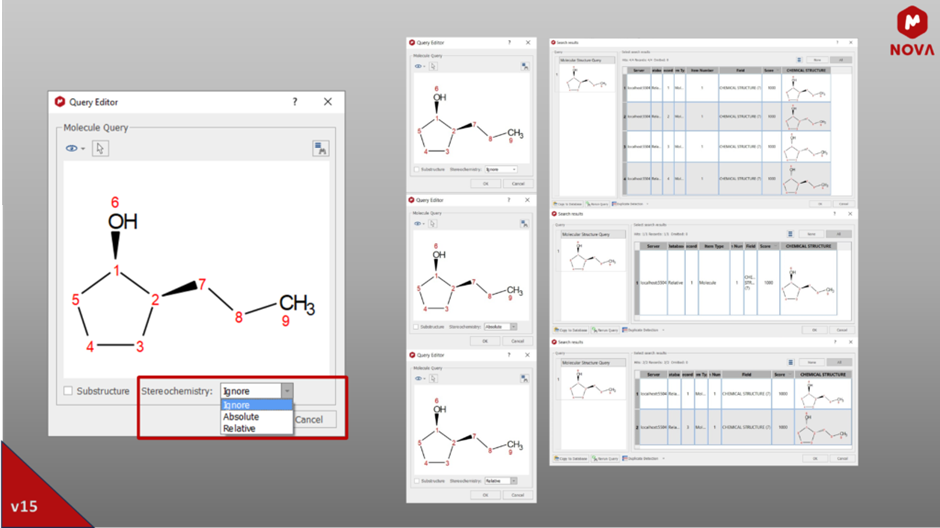
Rys. 10: Zapytanie o standardową cząsteczkę, na górze, zapytanie z opcją Absolute Stereochemistry, na środku i zapytanie z opcją Relative Sterochemistry, na dole.
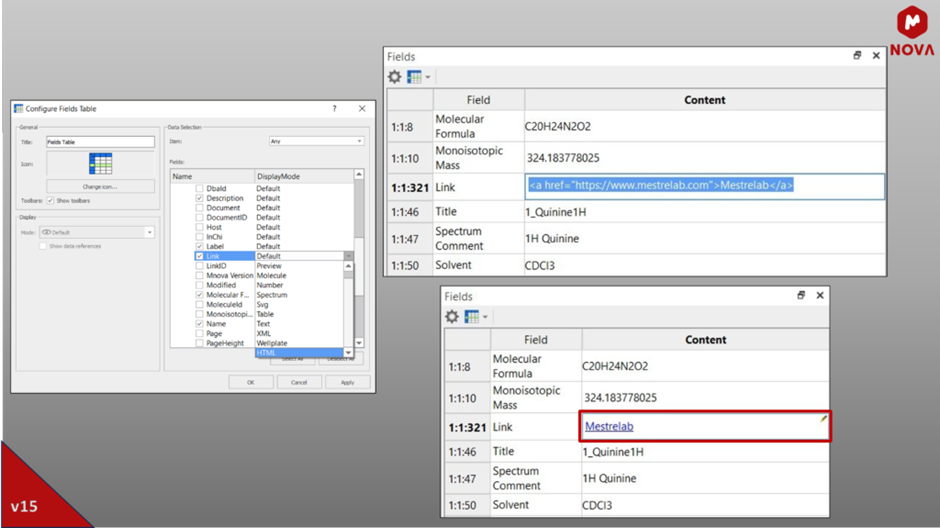
Rys. 11: Link dodany do witryny Mestrelab w tabeli Mnova DB.
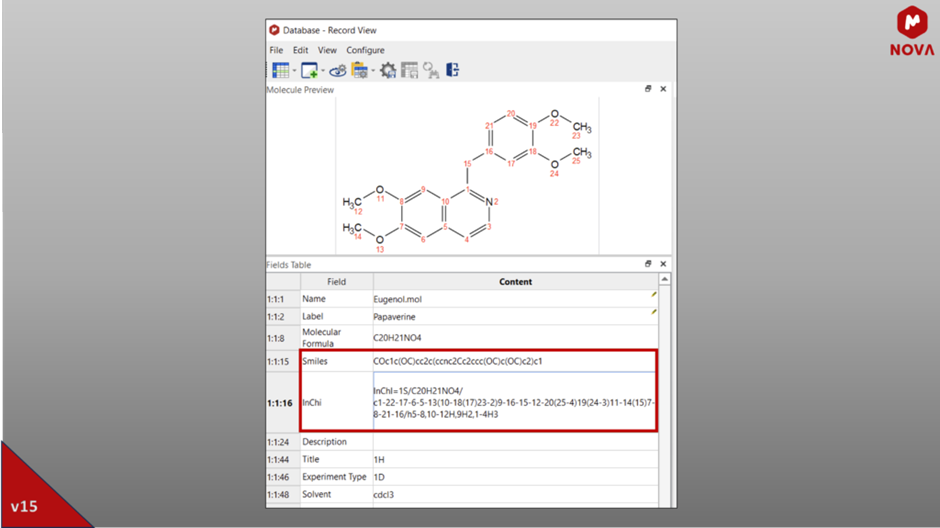
Rys. 12: Kolumny notacji SMILES i InChi wyświetlane w polach przeglądarki DB.
Nowości Mnova 14
Nowości funkconalne w programie Mnova 14